インフォメーション
【論文】新谷教授の論文がMicrobial Genomicsに掲載 2025/Sep
伝播するのは遺伝子だけじゃない——誤ラベルも。
年:2025(Published: 09 September 2025)
掲載先:Microbial Genomics(Letter / Volume 11, Issue 9)
著者:Masato Suzuki, Haruo Suzuki, Yosuke Nishimura, Hideaki Nojiri, Masaki Shintani
研究の要点
一言要約:プラスミド型別ツール 「MOB-typer」 が Pseudomonas の “IncP” 関連プラスミドを一括して誤分類しやすい問題を整理し,データベース上での誤ラベル拡散の実例と,その対処の方向性を示しました.
背景(なぜ重要か)
プラスミド(細菌が持つ可動性DNA)は,薬剤耐性(AMR)遺伝子などを水平伝播させる主要な要因です.そのため、流行解析(分子疫学研究では),「どのプラスミド群か」を正確に分類することが解析の前提となります.
何をしたか
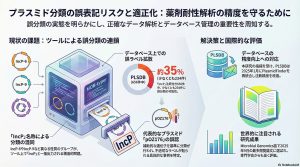
Pseudomonas における Inc 群の分類史(IncP-1〜P-14)と,腸内細菌科で用いられてきた IncP などの命名体系を整理し,"IncP” という名称が招く誤解・混乱されやすい点を明確化しました.そのうえで,MOB-typer およびその参照データベースが,IncP-6,IncP-7,IncP-9 など「IncP 由来の名称」をまとめて “IncP” と出力してしまう構造的問題を指摘しました.
何が分かったか(主要結果)
例として、PLSDB(プラスミドデータベース)で “IncP” と注釈された 656 件のうち,少なくとも 234 件は IncP-2,IncP-3,IncP-4,IncP-6,IncP-7,IncP-9,またはそれらのハイブリッドに相当し得ることを示しました.また,IncP-2 プラスミドの代表例である pOZ176 由来配列が,主要な複製開始遺伝子(RIP)ではなく補助的 RIP に相当するにもかかわらず “IncP” として扱われ,誤分類を誘発し得る点を具体例として示しました.
本研究での問題提起を受け,PLSDB 側では注釈精度向上のため,PlasmidFinder を追加で再統合(2025年1月)する対応が取られました.
価値・応用
AMR の分子疫学解析やメタゲノム解析において,誤ったプラスミドラベルが連鎖的に引用・再利用されるリスクを減らすために,ツールの併用や命名の扱いに関する実務的な注意点を共有します.「データベースをきれいに保つ」ための具体的な視点を提供する研究です.
本論文はMicrobial Genomics 誌において 2025年9月の「最も閲覧された論文の一つ」に選ばれました.
英国のMicrobiology Society の Chief Executive(Peter Cotgreave 博士)からも,本研究が研究コミュニティにとって価値ある成果として高く評価されているとのコメントが寄せられています.
本稿は、掲載論文(URL)に基づき生成AIを用いて要約・表現を作成しています。内容の正確性には配慮していますが、解釈や表現の過程で不正確な記載が含まれる可能性があります。正式な情報は原論文をご確認ください。